天府生命科技园园区企业今是科技与上海市生物医药技术研究院郑华军教授团队合作的学术成果 MtlA gene sequencing based on 3rd -generation sequencing for Vibrio typing 于2025年5月31日正式发表在 BMC Microbiology(IF=4)杂志(点击页面左下角“阅读原文”可查看文章全文)。

本文章构建了一项基于今是科技 G-seq500 纳米孔测序平台对mtlA基因进行全长测序的副溶血性弧菌(Vibrio parahaemolyticus)分子分型新方法。相较传统 MLST 方法,本方法极大简化了实验和分析流程,并提高了分型能力,这为食源性致病菌的快速分型提供了去中心化解决方案。
文库制备
通过一对带有标签序列的特异性引物对 33 例副溶血性弧菌样本的 mtlA 基因进行全长扩增(图一),扩增产物长度为 2085 bp。扩增产物混合纯化后,使用今是科技的样本制备通用试剂盒进行文库制备,再使用今是科技 G-seq500 纳米孔测序仪进行测序。

mtlA基因扩增引物设计示意图
数据质控
单张芯片单次测序产出了 199,658 条数据。经过对单一模版的多次重复测序数据(subreads)进行一致性矫正,下机数据的单序列读长众数为 2085 bp,单序列 Q 值众数为 35(图二)。
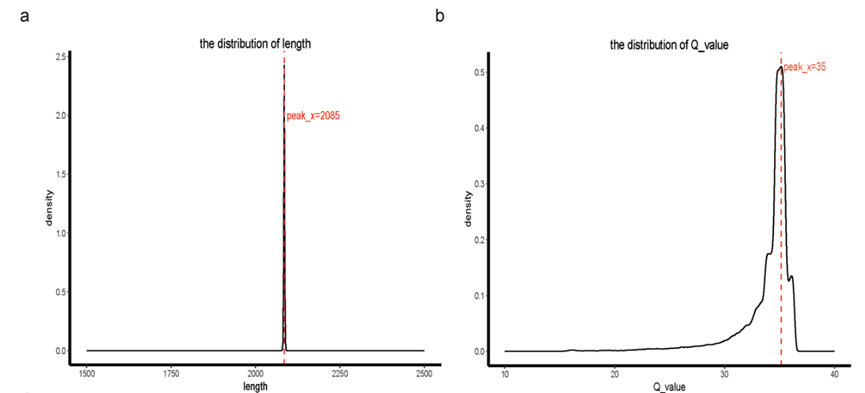
下机数据读长和Q值分布
数据分析
1. 使用二代测序结果作为参考基因组进行比对,今是纳米孔测序平台下机数据比对一致性平均为 99.88~99.93%,33 例样本中有 23 例的平均比对一致性大于等于 99.9%(表一)。这进一步验证了今是平台测序数据高度的准确性。
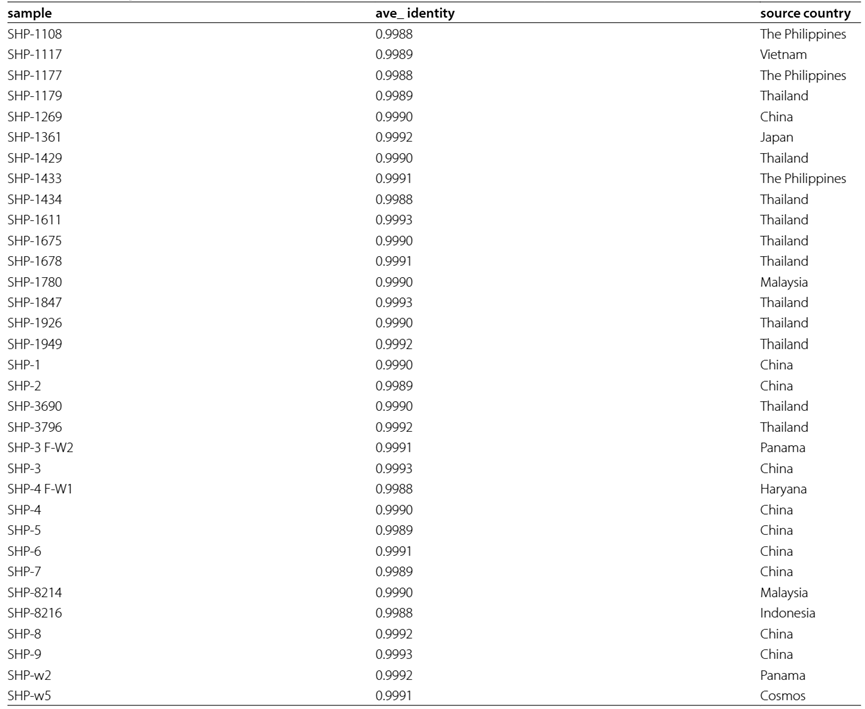
表一 下机单序列比对一致性
2. 分析结果显示,当目标区域(mtlA 基因)覆盖深度达到 4x 时就可以实现 100% 准确的序列判读(表二)。在本研究中,G-seq500 纳米孔测序仪单次测序产生了 64,086 条符合质控及拆分标准的下机序列,据此推算,单次测序就可以完成上万例样本的分型。
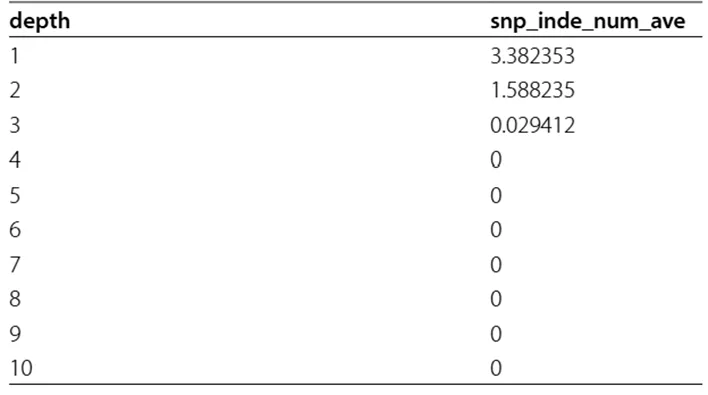
表二 不同覆盖深度下错误率
结论
本研究成功构建并验证了一种基于 mtlA 基因全长测序的副溶血性弧菌分子分型新方法。与传统的多位点序列分型(MLST)方法需要对多个基因进行扩增和测序不同,mtlA 分型只需对一个基因进行一步 PCR 反应即可完成,大大简化了实验流程。同时,mtlA 分型显示出更高的分辨率,并可以对 MLST 方法无法分型的菌株进行分型。
使用今是科技 G-seq500 纳米孔基因测序平台可以获得 >Q30 的 mtlA 基因全长测序数据,并能实现上万例样本同时测序,使得 mtlA 基因分型更加高效、便捷。今是科技 G-seq500 纳米孔测序仪具备低开机成本(芯片稳定复用10次)和实验流程简单高效的特性,使得这项技术有利于在地市级疾控、海关口岸及地方食品监控实验室等去中心化的场景下开展,极大加速全球范围内的菌株溯源和疫情监控,为保障公共卫生安全提供强有力的技术武器。
G-seq500测序流程
优势:
- 极简建库,最快 4 小时数据下机
- 测序芯片可室温存放,10次稳定复用
- 测序仪自动完成文库上机和芯片清洗


天府生命科技园微信